研究内容 | 早稲田大学大学院先進理工学研究科 生命医科学専攻 | 竹山研究室 生命分子工学研究室
- 1. 1細胞ゲノミクスを⽤いたマイクロバイオーム研究
- 2. 細胞レベルでの組織理解のための統合解析
- 3. ラマン分光法による細胞内分子探索技術の開発
- 4. 複合的データを解析するためのツール・アルゴリズム開発
- 5. 微生物の力を最大限活用した健康な土壌から環境にやさしい未来型農業を実現する
- 6. ムーンショット型研究開発事業における高校生との共同プロジェクト
- 7. 1細胞/微小組織マルチオミックスのオールインワン解析による生命科学研究の支援
- 8. マリンバイオテクノロジーと海洋環境モニタリング
目次
【技術 Technology】
【プロジェクト Project】
技術 Technology
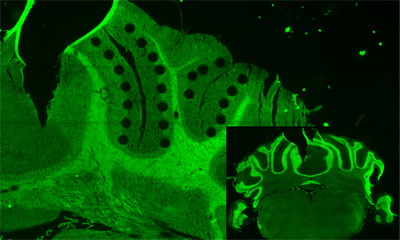
シングルセル 1細胞ゲノミクスを⽤いたマイクロバイオーム研究
海洋・土壌などのヒトを取り巻く環境から、ヒトの皮膚・腸内環境に至るまで、あらゆる箇所に微生物は存在します。環境微生物の多くは培養が難しく、その詳細解析には培養を経ずにゲノムを直接調べる方法が取られます。従来の環境微生物ゲノム解析では、様々な微生物ゲノムが混在した試料(メタゲノム)を取り扱っていたため、その中に埋もれた興味のある微生物のゲノムを取り出して再構築することは容易ではありませんでした。竹山研究室が開発したシングルセルゲノム解析法 SAG-gel1,2,3では、ピコリットル容量の微小な液滴の中に微生物を1細胞ずつ閉じ込めて個別に解析を行うことにより、たった1つの微生物細胞から高精度なゲノム情報を得ることができます。本技術を活用すれば、これまで誰も手にすることのなかった微生物ゲノムデータを次々に獲得することができます。本技術は、細菌やアーキアだけでなく、ウイルスのような更に小さな微生物も対象とできるため、環境中の様々な未知微生物のデータの蓄積を進めています。未知の微生物のゲノムデータからは、環境中における微生物の役割・他の微生物や宿主との相互作用を明らかにする手がかりが得られるだけでなく、産業・医療の発展に貢献する微生物由来の有用物質探索が可能です。「ムーンショット」や「マリンバイオテクノロジーと海洋環境モニタリング」をはじめ、様々なプロジェクトにおいて本技術が活用されています。
【参考論文】
- 1. Validation of the application of gel beads-based single-cell genome sequencing platform to soil and seawater
- 2. Single-cell genomics of uncultured bacteria reveals dietary fiber responders in the mouse gut microbiota
- 3. Targeted single-cell genomics reveals novel host adaptation strategies of the symbiotic bacteria Endozoicomonas in Acropora tenuis coral
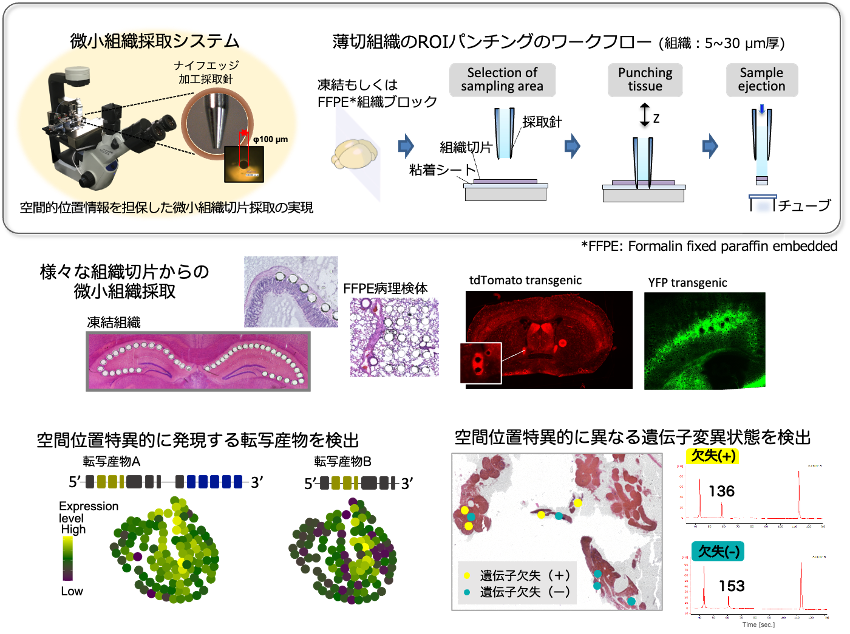
RNA-seq 細胞レベルでの組織理解のための統合解析
複雑な生命現象を理解するためには、ひとつひとつの細胞で働いている遺伝子の理解、即ち遺伝子発現解析が有効な手段になります。ゲノムシーケンス技術の目覚ましい発展により、発現する遺伝子の網羅的な配列解析が可能になりました。その一方で、細胞の組織内の空間的位置情報が細胞間ネットワークの理解に不可欠であり、生物学的機能の解明や、病因解明には必須であることがわかってきました。竹山研究室では、組織切片からマイクロオーダーの極微小領域を採取する微小組織採取技術1、凍結やFFPE*組織由来の1細胞もしくはそれに準ずる数十細胞を対象としたDNAやRNAを解析する技術2,3、およびそこから得られる配列情報をもとにした機能予測や組織形態との関連づけによる空間的トランスクリプトームを軸とした組織構成の解析技術4の開発を進めてきました。さらに、これらの技術を『生命科学・創薬研究支援基盤事業(BINDS)』において活用し、生体組織の機能理解や、医療分野への幅広い応用、さらに、細胞内に存在する遺伝子のみならず、タンパク質や代謝産物といったあらゆる生体分子の相互作用解析を実現する技術の構築を進めています。 (*FFPE: Formalin fixed paraffin embedded)
【参考論文】
- 1. Site-specific gene expression analysis using an automated tissue micro-dissection punching system
- 2. Effective microtissue RNA extraction coupled with Smart-seq2 for reproducible and robust spatial transcriptome analysis
- 3. Reproducible and sensitive micro-tissue RNA sequencing from formalin-fixed paraffin-embedded tissues for spatial gene expression analysis
- 4. Integrated spatial analysis of gene mutation and gene expression for understanding tumor diversity in formalin-fixed paraffin-embedded lung adenocarcinoma
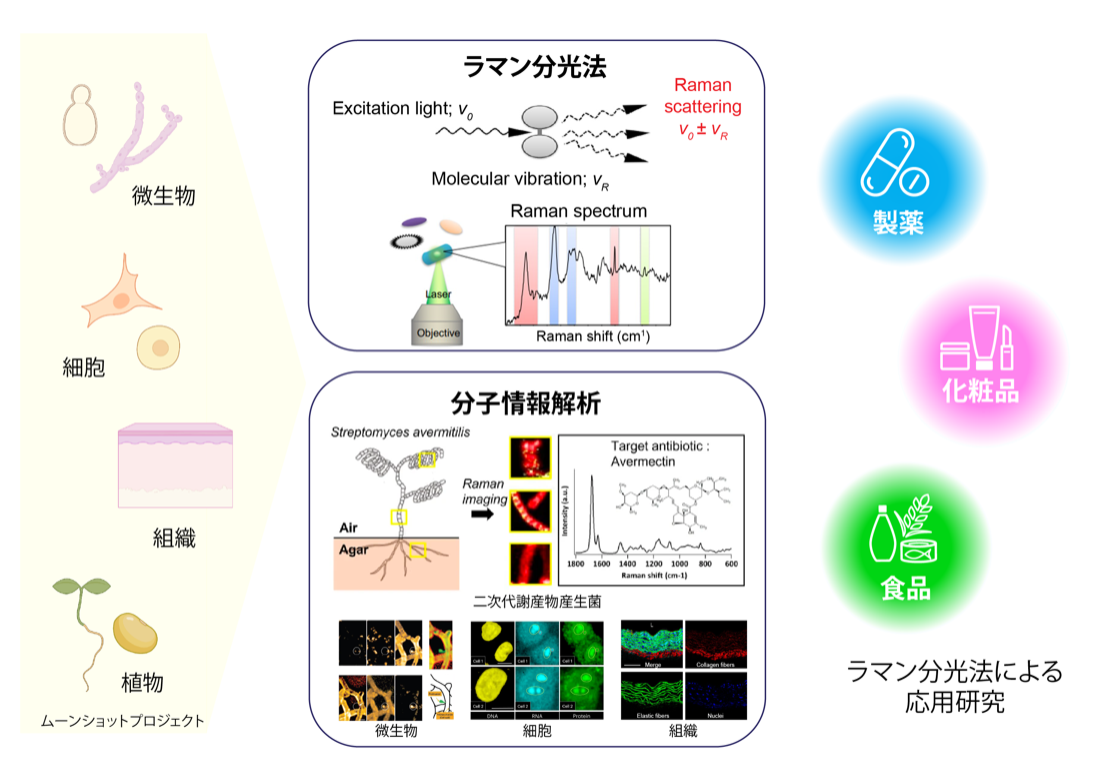
ラマン ラマン分光法による細胞内分子探索技術の開発
ラマン分光法は、1928年インドのC.V.ラマン博士によるラマン効果の発見に端を発する分光法で、ラベルフリー・低侵襲の分子解析技術です。歴史ある分光法ですが、ラマン散乱光の強度は微弱であるため、これを用いるためには様々な改良が必須でした。近年では、ハードウェアの技術進歩、情報解析技術を駆使したスペクトル解析手法の発展により、生体サンプルのような複雑な対象の中で、どの分子が、どのように分布しているかを解析できるようになってきました。竹山研究室では、産学連携による生体分子計測に適した装置・デバイス開発や、独自の解析アルゴリズムの構築を通じ、生体組織や細胞、微生物の中で種々の分子をラベルフリーで検出する技術を構築しています。微生物が産生する二次代謝産物を含め、各種生体分子ラマンスペクトルをライブラリー化することで、微生物1細胞レベルでの解析・スクリーニングができる技術の開発に取り組んでいます1,2,3。特に、ムーンショットプロジェクトにおいては、ダイズと共生する微生物のラマン分光解析を通して、宿主の生育に寄与する生理活性物質の探索、分子イメージング解析などを行っています。また、企業や他研究機関と実用応用を目指した共同研究も行っており、多岐にわたるラマン分光法の応用を目指しています。
【参考論文】
- 1. Mycelial differentiation linked avermectin production in Streptomyces avermitilis studied with Raman imaging
- 2. Raman Microspectroscopy Imaging Analysis of Extracellular Vesicles Biogenesis by Filamentous Fungus Penicilium chrysogenum
- 3. Single-cell metabolite detection and genomics reveals uncultivated talented producer
情報解析 複合的データを解析するためのツール・アルゴリズム開発
次世代シークエンサーの登場により、生物学を取り巻くデータは多種多様なものになりました。竹山研究室では、研究室内で開発した技術を用いて取得した複合的なビッグデータを、統合的に解析するためのツール・アルゴリズムの開発を行っています。これまでにロングリードシーケンサーによるシングルセルゲノムデータから完全な円形のゲノム配列を構築するscALA1(参照図左上)。シングルセルゲノムデータに含まれる環境中の汚染を取り除き、より高い精度にするSAG-QC2。複数のシングルセルゲノムデータを組み合わせることによって、完成度の高いゲノム情報を構築するccSAG3などを公開しました。また、極微小領域のRNA解析データを効率的に解析するための解析パイプライン4や、In vivoラマン分光法により取得したスペクトルから分子情報を抽出するためのプログラムを開発してきました。 さらに、開発したツール・アルゴリズムを、竹山研究室の全ての研究プロジェクトにおいて獲得した「メタゲノム」・「シングルセルゲノム」・「トランスクリプトーム」・「環境情報」などの複合的なビッグデータに応用することで、生物学的知見の創出を目指しています。独自のバイオテクノロジー技術と、独自の情報解析手法を組み合わせることで、様々な研究プロジェクトから最先端の知見を生み出せることを期待し研究を進めています。
【参考論文】
- 1. Revealing within-species diversity in uncultured human gut bacteria with single-cell long-read sequencing
- 2. SAG-QC: quality control of single amplified genome information by subtracting non-target sequences based on sequence compositions
- 3. Obtaining high-quality draft genomes from uncultured microbes by cleaning and co-assembly of single-cell amplified genomes
- 4. Single-cell metabolite detection and genomics reveals uncultivated talented producer
プロジェクト Project
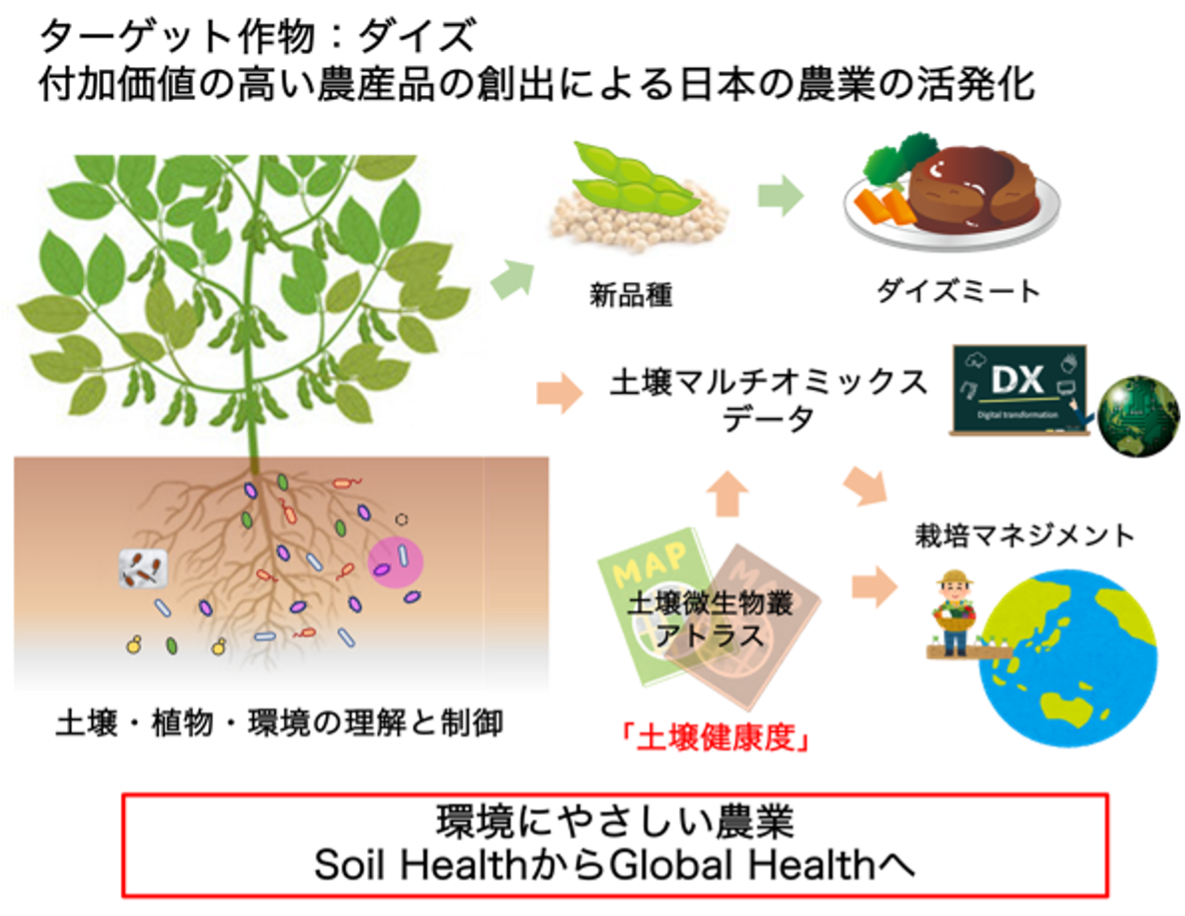
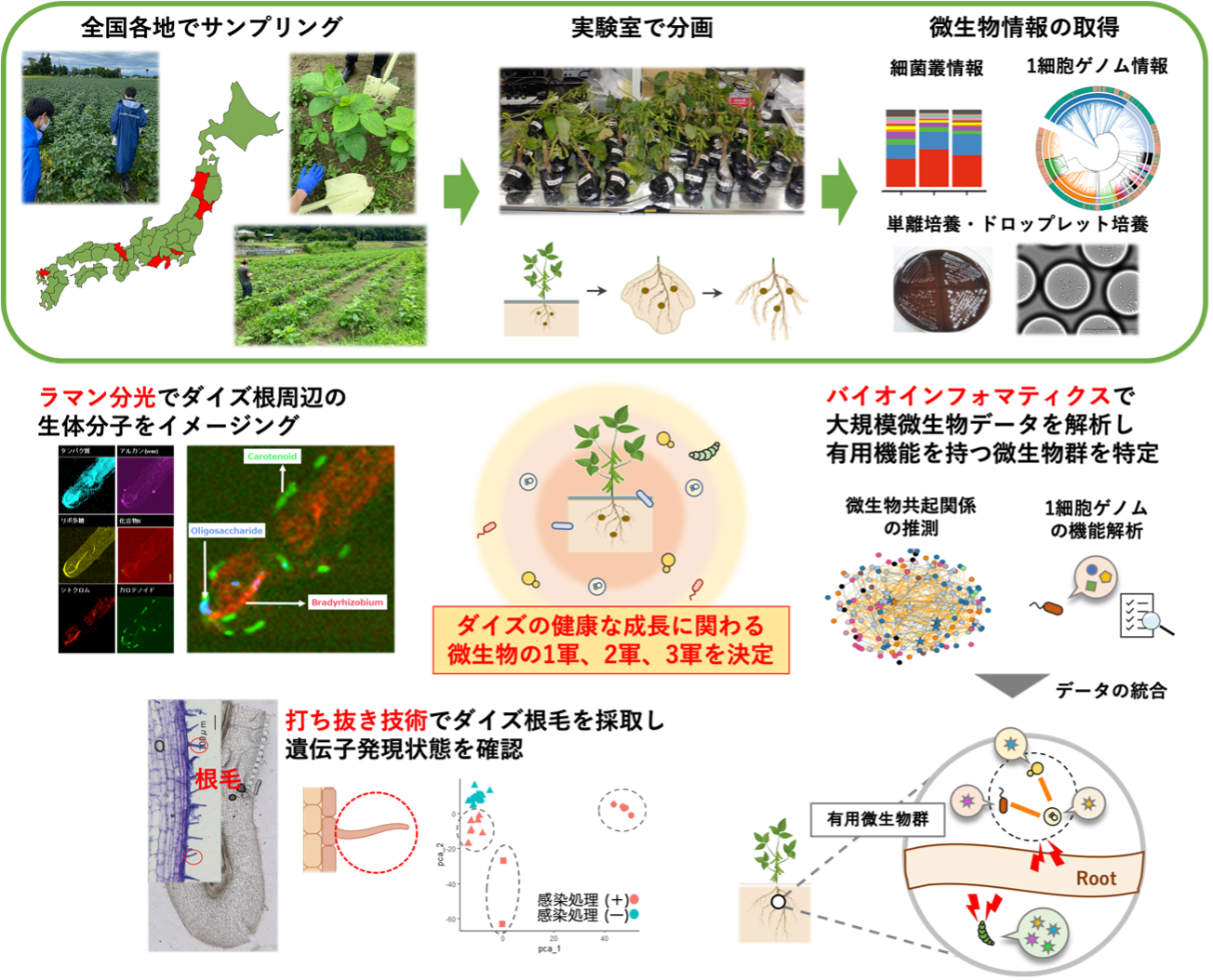
ムーンショット型研究開発事業 微生物の力を最大限活用した健康な土壌から環境にやさしい未来型農業の実現
ムーンショットとはその名の通り、本来は月に向かってロケットを打ち上げることを意味します。人類初の月面着陸を実現したアポロ計画から来ている言葉であり、ムーンショット型研究とは、「我が国発の破壊的イノベーションの創出を目指した、従来技術の延長にない、より大胆な発想に基づく挑戦的な研究開発」のことを指します。早稲田大学では、シングルセルゲノム解析技術や顕微ラマン分光法、空間オミックス解析といった最先端技術を用いて、多様な土壌に生息する微生物ゲノム情報の収集・解析を行い、それらをデータベース化した土壌微生物叢アトラスを作成しています。さらに本プロジェクトでは、他の研究機関の多数の研究者が構成する5つのサブグループが協力しており、作物、環境制御・測定、社会科学、栽培マネジメントなどのデータも取得しています。土壌・植物・環境の3つの要素を正確に把握し、それらの相互作用を理解・制御することを実現し、循環型協生農業を実現しようと研究を進めています。 竹山研究室ムーンショットプロジェクトの詳しい研究内容は以下のリンクから是非ご覧ください。
【竹山研究室ムーンショットプロジェクトHP】
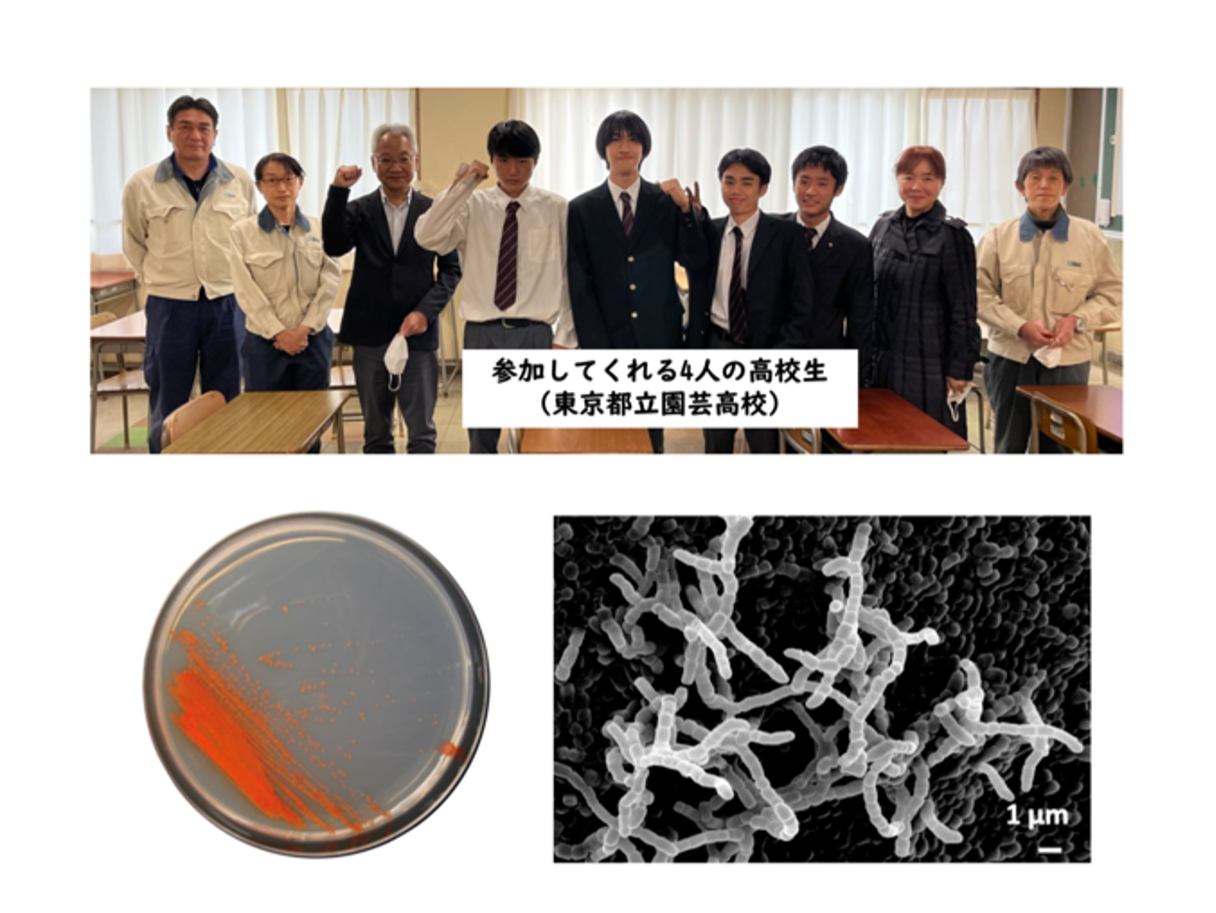
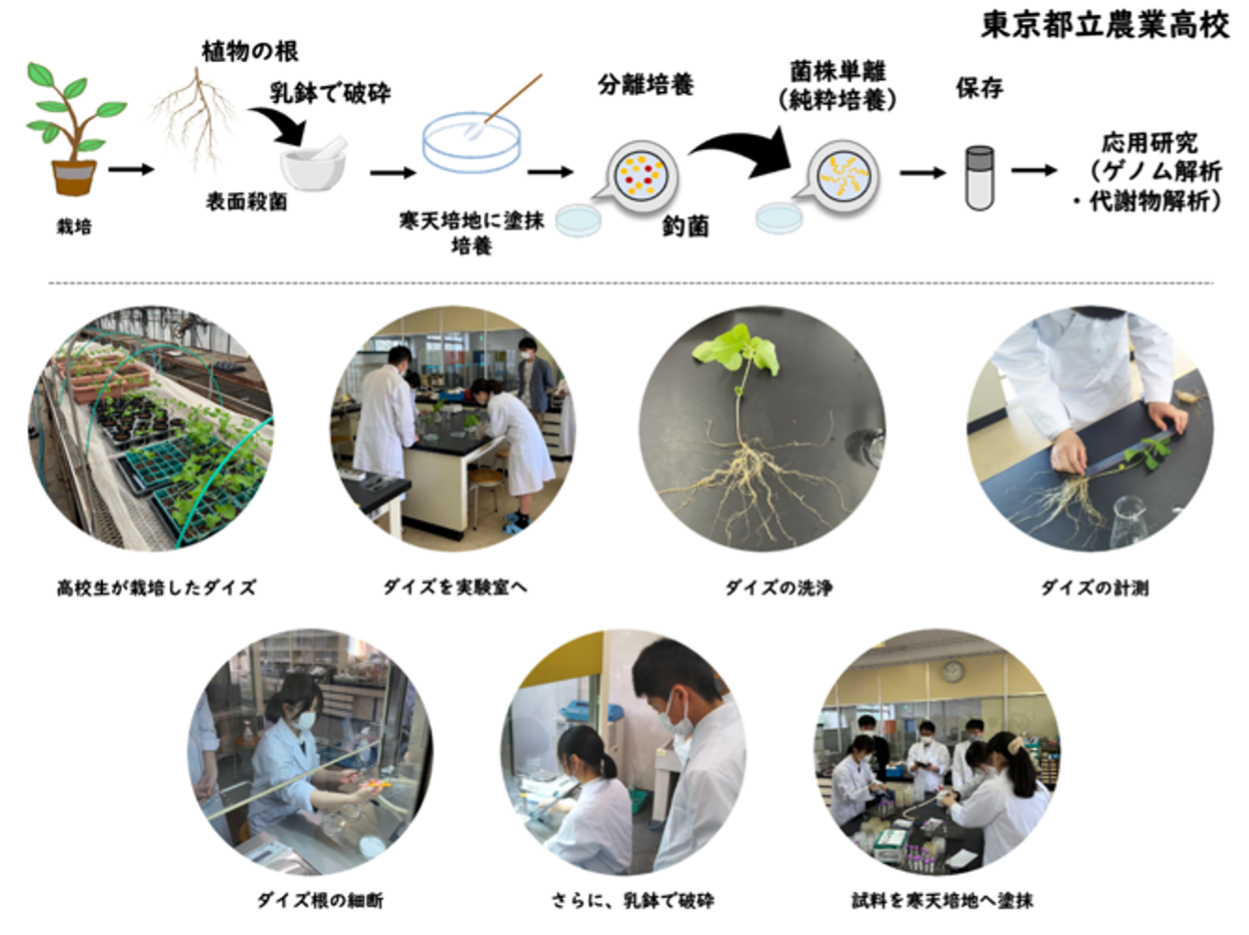
ムーンショット型研究開発事業 ムーンショット型研究開発事業における高校生との共同プロジェクト
マメ科植物の根には、こぶのようなものがあります。根粒菌が根に侵入して形成したもので 根粒と呼ばれています。根粒が形成されると、根粒菌は窒素固定を行うため、大豆にとって重要な働きをしています。植物の根には根粒菌の他に放線菌も生息しています。放線菌は原核微生物で、細胞の構造や大きさは他の細菌類と似ていますが、放射状に菌糸を伸ばし、様々な形の胞子を形成します。生態系においては、落葉などの有機物の分解や物質循環に関わる分解者として大きな役割を果たしています。放線菌は生物活性物質の生産者としても知られており、抗生物質など有用な天然物を生産します。竹山研究室のスタッフの指導のもと、高校生に放線菌の分離を行っています。東京都立農業高校と東京都立園芸高校の高校生が自ら育てた大豆を使って、根に生息する放線菌を分離しています。1株1株純粋培養した放線菌を観察し、多彩で多様なコロニーに触れています。これから将来を担う高校生が、身の回りに生息する微生物を感じ、興味を持ってくれることを期待します。
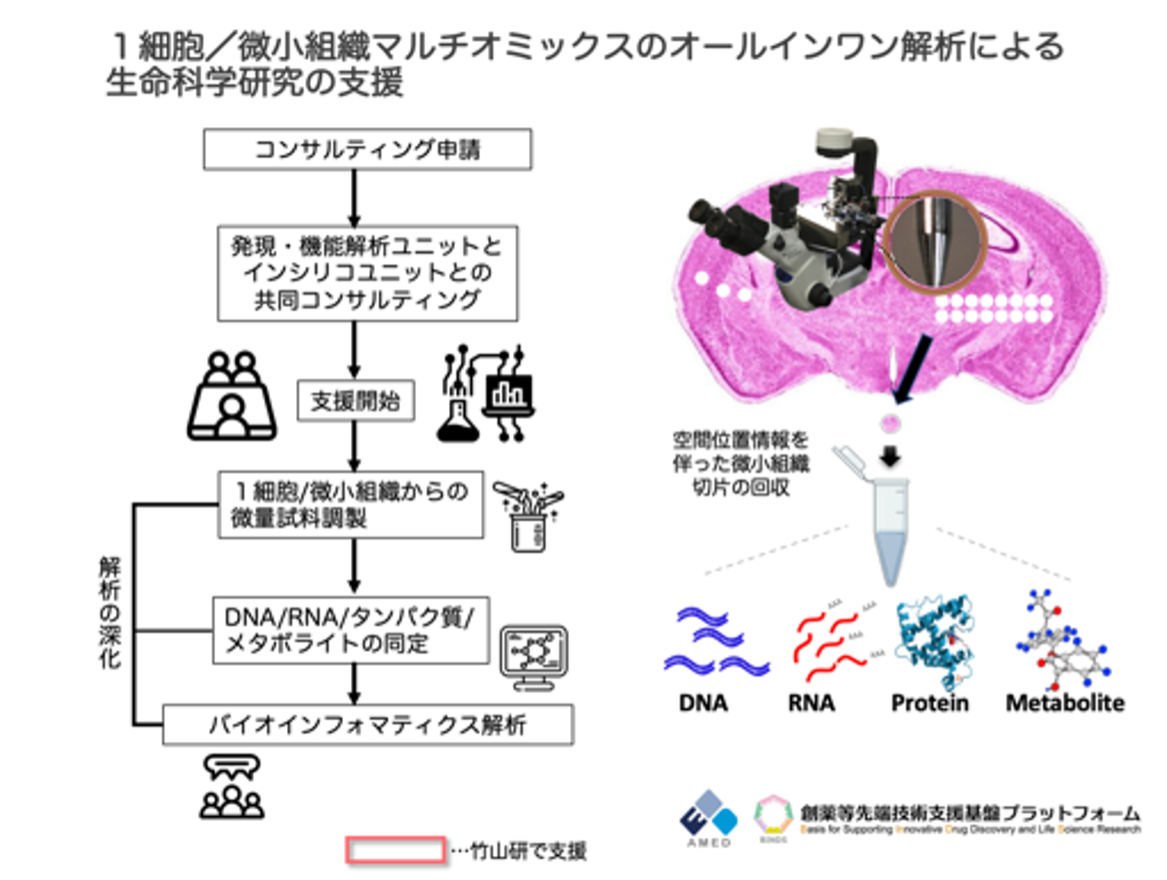
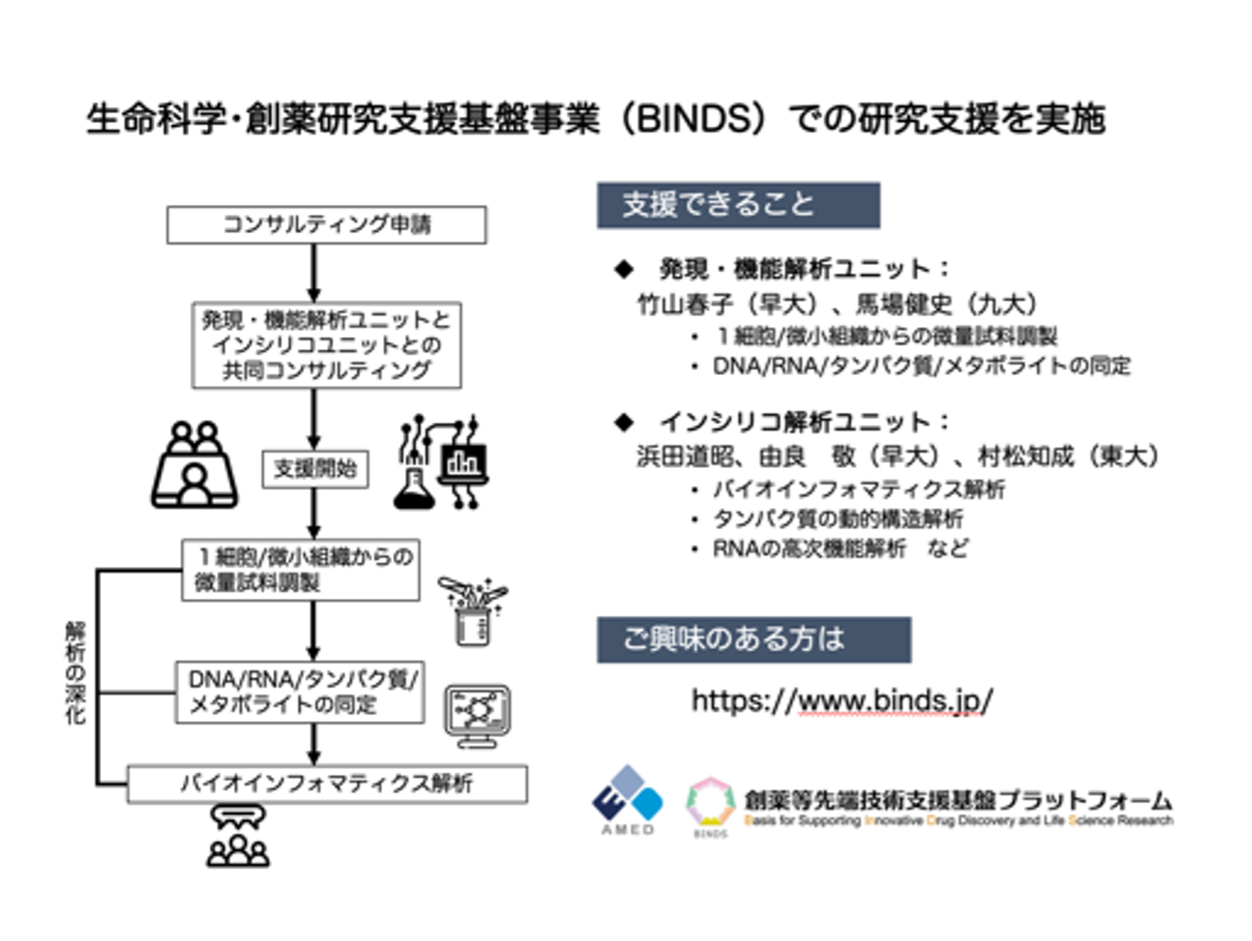
生命科学・創薬研究支援基盤事業(AMED_BINDS) 1細胞/微小組織マルチオミックスのオールインワン解析による生命科学研究の支援
竹山研究室では、小さな組織や1つの細胞に対してDNA、RNA、タンパク質、代謝産物の解析およびバイオインフォマティクス解析を包括的に活用するプロジェクトに参加しています。竹山研究室が持つ、組織内の微小空間の遺伝子発現を解析できる技術を応用し、微小組織や1つの細胞からのRNA解析にとどまらず、タンパク質や代謝産物の解析を行っています。さらに、従来からの課題であるウエット(実験)とドライ(解析)の連携を強化する体制をとっています。 これまでの支援実績は、生命現象の根幹に迫る分野や、診断応用に関する分野など多岐にわたっています。例えば、分裂酵母細胞が休眠から目覚める際の遺伝子の発現状態変化の解析1、微小な脳領域における不安障害の機能的バイオマーカー探索2や、劇症型溶血性レンサ球菌感染症における新規骨髄系細胞の遺伝子発現解析3、腫瘍内組織不均一性解析に基づくHPV18型の発癌機構と分化調整機構の検討4などがあります。
【参考論文】
- 1. Time-lapse single-cell transcriptomics reveals modulation of histone H3 for dormancy breaking in fission yeast
- 2. Distinctive Regulation of Emotional Behaviors and Fear-Related Gene Expression Responses in Two Extended Amygdala Subnuclei With Similar Molecular Profiles
- 3. Sequential Sensing by TLR2 and Mincle Directs Immature Myeloid Cells to Protect against Invasive Group A Streptococcal Infection in Mice
- 4. Cells with stem-like properties are associated with the development of HPV18-positive cervical cancer
マリンバイオテクノロジーと海洋環境モニタリング
海洋は地球表面の約70%を占め、多様な生物が生存している重要な環境です。生物の持つゲノム情報は、その生物や生育環境のことを詳しく調べることができるだけでなく、バイオテクノロジーにより環境問題の解決や人類の生活向上に役立つことが期待されます。竹山研究室では、環境オミックス技術とシングルセルゲノム技術という最先端の手法を使って、海洋での遺伝情報を大規模に収集し、蓄積しています。収集した遺伝情報の利用例として、これまでに海洋の季節変動や深海を含む様々な環境適応に関する、海洋微生物やウイルス、遺伝子の探索を進めており、新たな有用物質の開拓や地球温暖化などの環境問題の予測や解決に向けた手掛かりになることを期待しています。これまでに、海外の研究機関とも協力し、サンゴ・カイメンなどの共生細菌や海水中の微生物に関する研究を行ってきました1,2,3。 現在、より効率的な次世代の水産資源や海洋環境モニタリング手法の開発を目指して、音響探査技術とゲノム解析を組み合わせた調査・研究を実施しています。現在は、主に駿河湾を対象としてフィールド調査を継続して行なっており、現地で収集したデータをこれらの研究に活用しています。竹山研究室では、海洋環境の研究に用いることができる様々な技術を開発してきました。これらの技術と実際の海洋環境調査を組み合わせることで、地球環境問題解決や人類の生活向上に向けた先進的な研究を行っています。
【参考論文】
- 1. An environmental bacterial taxon with a large and distinct metabolic repertoire
- 2. The Effect of Co-Culture of Two Coral Species on Their Bacterial Composition Under Captive Environments
- 3. Targeted single-cell genomics reveals novel host adaptation strategies of the symbiotic bacteria Endozoicomonas in Acropora tenuis coral